A01-5 機動性ゲノムの認識機構の実体と種特異的応答の解明

研究代表者
眞貝 洋一 Shinkai Yoichi
理化学研究所 開拓研究本部 主任研究員
- 研究室https://shinkai.riken.jp/
- researchmaphttps://researchmap.jp/read0057299

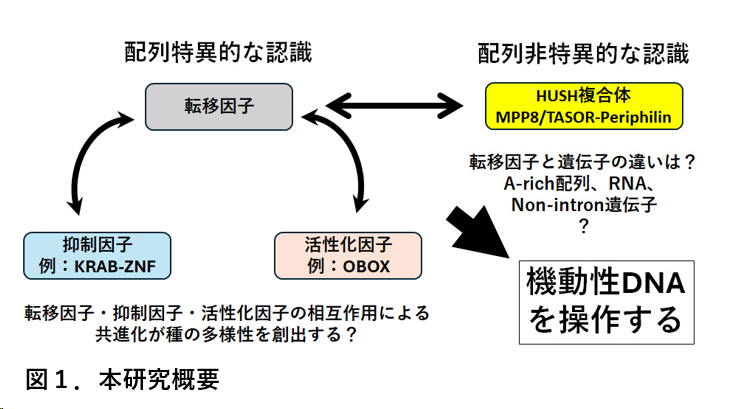
生命(宿主)が機動性ゲノムをその制御下に置くには、まずそれを識別することが重要である。機動性ゲノムの識別には、塩基配列特異的なものと、それ以外のものがあることが示されている。本課題では、この2つの識別・認識機構のさらなる解明を目指す。
1.配列特異的認識機構の実体の解明と進化的考察:宿主と機動性ゲノムは認識に関しても互いにせめぎ合いながら進化してきた。この共進化は生物種間のゲノム多様性の創出に加え、種特異的な遺伝子制御ネットワークの確立にも貢献し、種特異的形質や疾患形成に於いて重要な役割を担ってきたと考えられているが、その分子的実体はほとんど明らかでない。そこで、機動性ゲノムの認識を規定する配列と認識因子を多生物種で包括的に明らかにする手法を構築し、情報・遺伝学的解析により、この課題の解決に取り組む。まず、機能的スクリーニングで得られたゲノム情報を機械学習や遺伝系統解析にかけ、機動性ゲノムの認識配列を抽出するとともに、機動性ゲノムが宿主の標的から逃れる過程でどのように配列を変化させてきたかを明らかにする。さらに、機動性ゲノムの多様化と関連して多様化したと推測されるKRAB-ZNFファミリー遺伝子とHomeoboxファミリー遺伝子の発現ベクターライブラリーを作成し、その認識配列を網羅的に調べる実験系を確立する。両者の結果を統合して、進化における機動性ゲノムとその認識因子の多様化とせめぎ合いの分子機構を明らかにする。
2.配列以外の認識機構のメカニズムの解明:近年、The human silencing hub (HUSH)と呼ばれる複合体が機動性ゲノムの転写抑制に寄与すすること、さらに特異的な塩基配列ではなくA-richで長いイントロンレスの遺伝子を標的とし、転写抑制することが示された。しかし、今のところ、その認識の詳細な機構は明らかでない。そこで、HUSH複合体が、どのように機動性ゲノムを識別しているのか、そのためにHUSH複合体を有するが進化的に離れた生物間での機能を検討したり、HUSH複合体を持たない生物種にHUSH複合体を導入するとどのようなことが起きるのかを解析、さらに有田計画研究代表と構造学的解析も進めながら、この認識と抑制機構の実体を明らかにする。
研究業績 ※責任著者
眞貝 洋一
- ※Fukuda K, et al., ※Shinkai Y.
Epigenetic plasticity safeguards heterochromatin configuration in mammals.
Nucleic Acids Res. gkad387. (2023) - ※Fukuda K, et al., ※Shinkai Y.
Potential role of KRAB-ZFP binding and transcriptional states on DNA methylation of retroelements in human male germ cells.
eLife 11:e76822. (2022) - ※Fukuda K, et al., ※Shinkai Y.
Regulation of mammalian 3D genome organization and histone H3K9 dimethylation by H3K9 methyltransferases.
Communications Biology 4(1):571. (2021) - Fukuda K, et al., ※Shinkai Y.
A CRISPR Knockout Screen Identifies SETDB1-target Retroelement Silencing Factors in Embryonic Stem Cells.
Genome Res. 28(6):846-858. (2018) - ※Kato M, et al., ※Shinkai Y.
Evidence for a general role of the histone methyltransferase Setdb1 in endogenous retrovirus silencing, not limited to early embryonic cells.
Nat Commun. 9(1):1683. (2018)
福田 渓
- ※Fukuda K, et al., ※Shinkai Y.
Epigenetic plasticity safeguards heterochromatin configuration in mammals.
Nucleic Acids Res. gkad387. (2023) - ※Fukuda K, et al., ※Shinkai Y.
Potential role of KRAB-ZFP binding and transcriptional states on DNA methylation of retroelements in human male germ cells.
eLife 11:e76822. (2022) - ※Fukuda K, et al., ※Shinkai Y.
Regulation of mammalian 3D genome organization and histone H3K9 dimethylation by H3K9 methyltransferases.
Communications Biology 4(1):571. (2021) - Fukuda K, et al., ※Shinkai Y.
A CRISPR Knockout Screen Identifies SETDB1-target Retroelement Silencing Factors in Embryonic Stem Cells.
Genome Res. 28(6):846-858. (2018) - Fukuda K, et al., ※Sasaki H.
Evolution of the sperm methylome of primates is associated with retrotransposon insertions and genome instability.
Hum Mol Genet 26, 3508-3519. (2017)
