A02-4 機動性DNAエレメントによるゲノム再編成と表現型多様化・種分化促進機構の解明

研究代表者
菊地 泰生 Kikuchi Taisei
東京大学 大学院新領域創成科学研究科 多細胞生物システム学分野 教授

研究分担者
杉本 亜砂子 Sugimoto Asako
東北大学 大学院生命科学研究科 脳生命統御科学専攻 発生ダイナミクス分野 教授

研究分担者
春田 奈美 Haruta Nami
東北大学 大学院生命科学研究科 脳生命統御科学専攻 発生ダイナミクス分野 助教
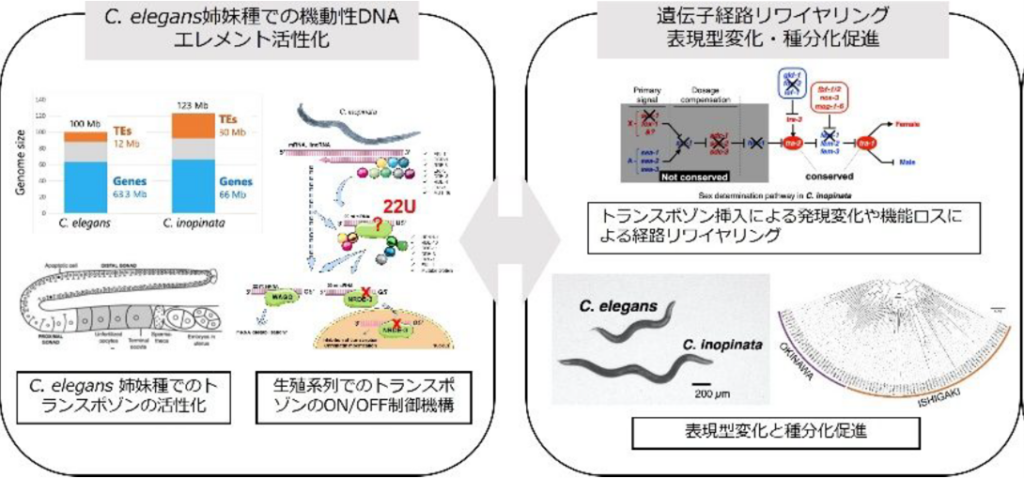
トランスポゾンなどの機動性DNAエレメントは、その増幅や転移によりゲノム構造を変化させ、結果として表現型多様化と種分化を促進していると考えられるが、その具体的なプロセスについては実験的な検証が困難なため解明が進んでいない。モデル線虫として広く用いられてきたC. elegansではトランスポゾンの転移が抑制されているが、申請者らはC. elegans姉妹種(C. inopinata)でトランスポゾンが増幅し、高い転移活性を持っていることを見出した。石垣島で発見されたC. inopinataは、最近縁種であるにもかかわらず、体がC. elegansの2倍以上巨大化しており、生態や生殖様式もC. elegansと大きく異なる。申請者らのゲノム比較と遺伝子機能解析により、C. inopinataではトランスポゾンの挿入により、small RNA代謝や性決定などに関わる遺伝子の一部が破壊されており、様々な遺伝子経路のリワイヤリングが起きていることが明らかとなってきた。また、南西諸島の野生株の解析により、C. inopinataにおいて島間でゲノムの構造多様化と部分的生殖不和合が存在していることが明らかとなった。本研究では、Caenorhabditis線虫(C. elegansとC. inopinata)をモデル系とし、高精度ゲノム構造解析(菊地)、遺伝子操作による表現型解析(杉本・春田)を統合的に用いることで、機動性DNAエレメントによる表現型変化促進や種分化についての実験的な検証を行う。具体的には、線虫種間・亜種間比較により1)small RNAパスウェイやヒストン修飾等による生殖系列での機動性DNAエレメントのON/OFF制御機構とその進化過程、2)機動性DNAエレメントによる遺伝子経路リワイヤリングと表現型多様化、3)機動性DNAエレメントによるゲノム再編成と種分化へ寄与の理解を目指す。
研究業績 ※責任著者
菊地 泰生
- Lee YC, et al., Kikuchi T.
Single-worm long-read sequencing reveals genome diversity in free-living nematodes.
Nucleic Acids Research 51(15): 8035-8047. (2023) - Kounosu A, et al., Sugimoto A, ※Kikuchi T.
Syntenic relationship of chromosomes in Strongyloides species and Rhabditophanes diutinus based on the chromosomelevel genome assemblies.
Philosophical Transactions of the Royal Society B. 15(379). (2023) - Yoshida K, et al., Kikuchi T.
Chromosome fusions repatterned recombination rate and facilitated reproductive isolation during Pristionchus nematode speciation.
Nature Ecology & Evolution, 1-16. (2023) - ※Kikuchi T, et al.
Genome of the fatal tapeworm Sparganum proliferum uncovers mechanisms for cryptic life cycle and aberrant larval proliferation.
Commun. Biology, 4, 649 (2021) - International Helminth Genomes Consortium (コンソーシアム)
Comparative genomics of the major parasitic worms.
Nature Genetics, 51(1), 163 (2019)
杉本 亜砂子
- ※Tamagawa K, et al., Kikuchi T, Haruta N, ※Sugimoto A, ※Makino T.
Evolutionary changes of noncoding elements associated with transition of sexual mode in Caenorhabditis nematodes
Science Advances, 10(37), eadn9913 (2024) - Hatanaka A, Tamagawa N, Haruta N, ※Sugimoto A.
The impact of differential transposition activities of autonomous and nonautonomous hAT transposable elements on genome architecture and gene expression in Caenorhabditis inopinata.
Genetics 227 (2), iyae052 (2024) - Kawahara K, et al., Kikuchi T, Sugimoto A, ※Kawata M.
Differentially Expressed Genes Associated with Body Size Changes and Transposable Element Insertions between Caenorhabditis elegans and Its Sister Species, Caenorhabditis inopinata
Genome Biology and Evolution, 15 (4) evad063 (2023) - ※Kanzaki K, et al.,※Sugimoto A, ※Kikuchi T.
Biology and genome of a newly discovered sibling species of Caenorhabdiits elegans
Nature Communications 9 (1) 3216 (2018) - Namai S, ※Sugimoto A.
Transgenesis by microparticle bombardment for live imaging of fluorescent proteins in Pristionchus pacificus germline and early embryos
Development Genes and Evolution 228, 75-82 (2018)
春田 奈美
- ※Tamagawa K, et al., Kikuchi T, Haruta N, ※Sugimoto A, ※Makino T
Evolutionary changes of noncoding elements associated with transition of sexual mode in Caenorhabditis nematodes
Science Advances, 10(37), eadn9913 (2024) - Hatanaka A, Tamagawa N, Haruta N, ※Sugimoto A.
The impact of differential transposition activities of autonomous and nonautonomous hAT transposable elements on genome architecture and gene expression in Caenorhabditis inopinata.
Genetics 227 (2), iyae052 (2024) - ※Haruta,N, et al., ※Sugimoto, A.
A germline-specific role for unconventional components of the g-tubulin complex in Caenorhabditis elegans.
J Cell Sci. (2023) 136(13):jcs260922. - Oomura, S, et al., Haruta, N, ※Sugimoto A.
Transgenesis of the gonochoristic nematode Caenorhabditis inopinata by microparticle bombardment with hygromycin B selection.
MicroPub Bio, (2022)
doi: 10.17912/micropub.biology.000564.
