A01-4 機動性DNAエレメントの発動・抑制の構造基盤とその制御技術の開発


研究分担者
西山 敦哉 Nishiyama Atsuya
東京大学 医科学研究所 癌・細胞増殖部門 准教授
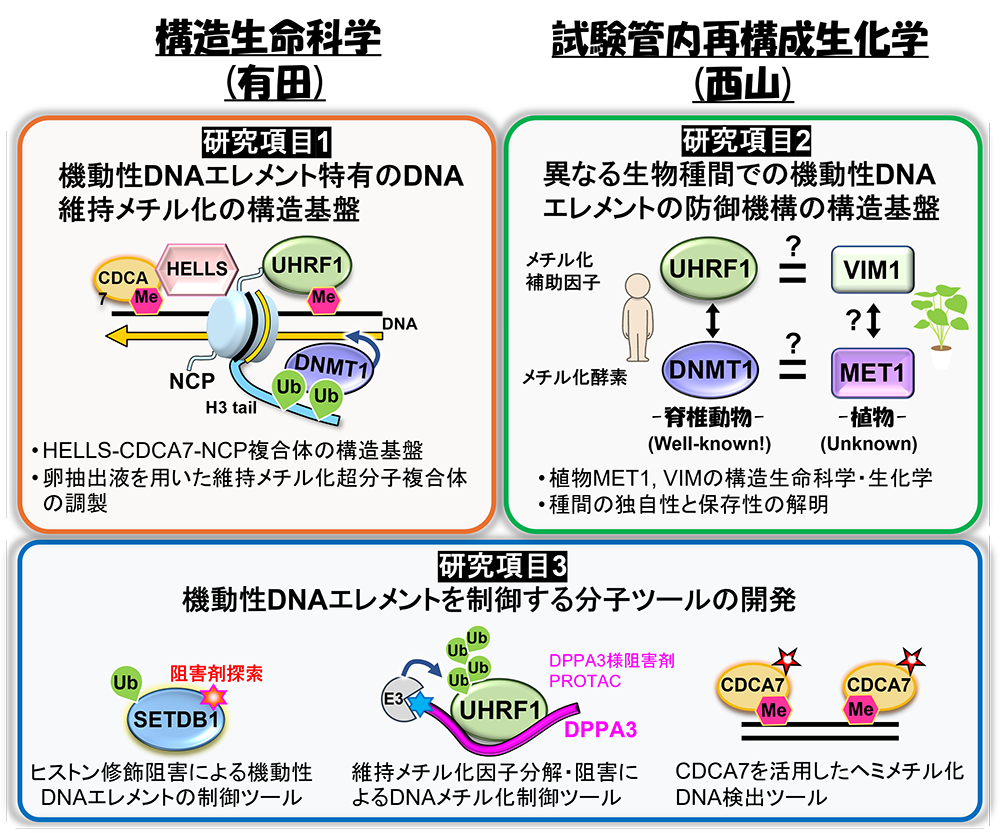
当計画班は、クロマチンリモデラーHELLSの制御因子であるCDCA7が、クロマチン上のヘミメチル化DNAを認識し、機動性DNAエレメントに特有のDNA維持メチル化を担うことを明らかにした。今後、CDCA7/HELLSとヌクレオソームの複合体をツメガエル卵抽出液を用いてin vitroで再構成し、機動性DNAエレメント上に形成される維持メチル化超分子複合体の構造を、クライオ電子顕微鏡単粒子解析により解明することを目指す。また、哺乳類の維持メチル化因子DNMT1およびUHRF1の植物ホモログであるMET1およびVIM1についても、クライオ電子顕微鏡による構造解析を進め、植物におけるDNA維持メチル化の構造基盤を解明する(連携:藤班)。これにより、種を超えて保存された機動性DNAエレメントの発動抑制機構の分子基盤に迫る。さらに、得られた立体構造情報に基づき、機動性DNAエレメントの発動と防御を制御する分子ツールの開発にも取り組む。具体的には、CDCA7のヘミメチル化DNA結合を特異的に阻害するモノボディーの開発(連携:林班)、UHRF1阻害因子DPPA3の構造情報をもとにしたPROTACやペプチド阻害剤の設計、配列特異的なDNAメチル化制御を可能にする改変型MET1の構築を進める。
また、内因性レトロエレメントの抑制に重要なヒストンメチル化酵素SETDB1については、その活性化機構を構造生物学的に解明し、得られた構造情報をもとに機能阻害剤の探索を行う(連携:眞貝班)。
研究業績 ※責任著者
有田 恭平
- Hata K, et al., ※Arita K.
Structural basis for the unique multifaceted interaction of DPPA3 with the UHRF1 PHD finger.
NAR. 28 12527-12542 (2022) - Kikuchi A, et al., ※Arita K.
Structural basis for activation of DNMT1.
Nature Communications 13, 7130 (2022) - Funyu T, et al., ※Arita K.
Preparation of the ubiquitination-triggered active form of SETDB1 in E. coli for biochemical and structural analyses.
The Journal of Biochemistry 170 655-662 (2021) - Kori S, et al., ※Arita K.
Serine 298 phosphorylation in linker 2 of UHRF1 regulates ligand-binding property of its tandem Tudor domain.
J. Mol. Biol. 432 4061-4075, 2020. - Kori S, et al., ※Arita K.
Structure of the UHRF1 Tandem Tudor Domain Bound to a Methylated Non-histone Protein, LIG1, Reveals Rules for Binding and Regulation.
Structure 27 485-496 (2019)
西山 敦哉
- Wassing IE, et al., ※Nishiyama A.
CDCA7 is an evolutionarily conserved hemimethylated DNA sensor in eukaryotes.
Science Advances 10(34):eadp5753. 2024. - Miyashita R, et al., ※Nishiyama A.
The termination of UHRF1-dependent PAF15 ubiquitin signaling is regulated by USP7 and ATAD5.
Elife, 12:e79013 2023. - Kumamoto S, et al., ※Nishiyama A.
Structural basis for the unique multifaceted interaction of DPPA3 with the UHRF1 PHD finger.
Nucleic Acids Res. 28 12527-12542, 2022. - ※Nishiyama A, et al.,
Two distinct modes of DNMT1 recruitment ensure stable maintenance DNA methylation.
Nature Commun.11, 1222 (2020) - Ishiyama S, et al., Nishiyama A.
Structure of the Dnmt1 reader module complexed with a unique two-mono-ubiquitin mark on histone H3 reveals the basis for DNA methylation maintenance.
Mol Cell 68, 350-360 (2017) - ※Nishiyama A, et al., Nakanishi M.
Uhrf1-dependent ubiquitylation of histone H3 at lysine 23 couples maintenance DNA methylation and DNA replication.
Nature 502, 249-253 (2013)
