A02-2 機動性DNAエレメントが駆動する生殖サイクル

研究代表者
岩崎 由香 Iwasaki Yuka
理化学研究所 生命医科学研究センター 非コードゲノム機能研究チーム チームディレクター

研究分担者
坂下 陽彦 Sakashita Akihiko
理化学研究所 バイオリソース研究センター ゲノム可塑性発生制御研究チーム チームディレクター
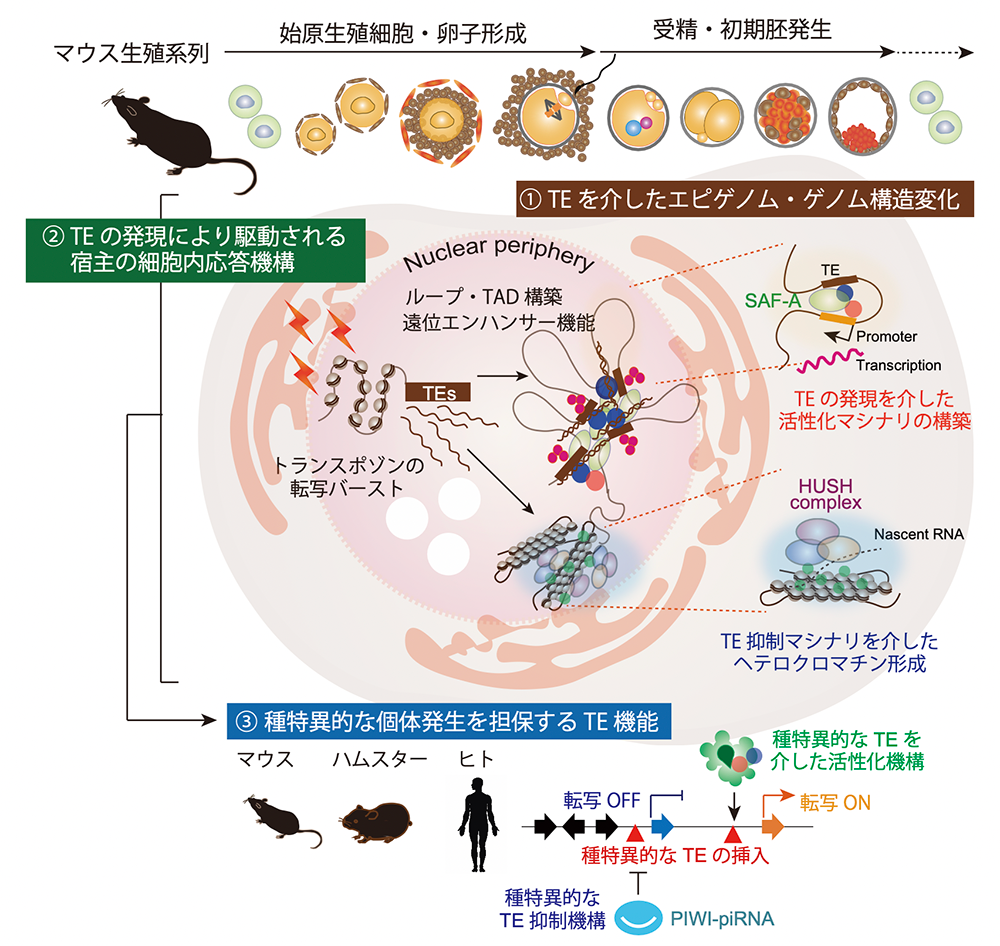
本研究では、配偶子形成(岩崎)と初期発生(坂下)といった生殖サイクルの各ステージにおいて、機動性DNAエレメントの機能的役割を評価し、世代を超えたトランスポゾンの動態や機能を理解する。さらに、マウス・ハムスター・ヒトといった複数の生物種を対象とした解析を行うことで、種特異的なトランスポゾンの個体発生への寄与を明らかにする。岩崎は、機動性DNAエレメントの発現とその暴走抑止機構が互いに戦うことで生まれる相克が、生殖サイクルのとくに卵子形成時に有する機能的役割を理解する。また坂下は、受精卵・初期発生胚において生じる機動性DNAエレメントの大規模な転写惹起に焦点を当て、宿主ゲノムのクロマチン再構築制御ならびに細胞運命決定機構におけるこれらの機能的意義を明らかにする。さらに興味深いことに、生殖系列細胞では、自然免疫応答機構として知られる外来性ウイルスの構成因子(核酸やタンパク質)に対する一連の細胞質センサーが活性化していることが多くの先行研究によって示唆されている。これらの機構がトランスポゾンに対する宿主細胞の逐次的な応答機構の一部として転用されている可能性が強く想定されるため、トランスポゾンの発現により誘導される生殖系列特異的な宿主応答機構の全貌も明らかにする。領域内共同研究をベースに、生殖系列におけるトランスポゾンの発現と宿主の応答機構、その結果として生じる宿主ゲノムの高次クロマチン変化や遺伝子発現変化、さらには種固有のトランスポゾン配列をゲノム上に多く内包する多様な生物種の比較解析を通して最終的な表現型へのインパクトを体系的に解明する。
研究業績 ※責任著者
岩崎 由香
- Ariura M, et al.,Iwasaki YW.
Drosophila Piwi distinguishes transposons from mRNAs by piRNA complementarity and abundance.
Cell Rep, 43: 115020 (2024) - Takeuchi C, et al., ※Iwasaki YW.
Mod(mdg4) variants repress telomeric retrotransposon HeT-A by blocking subtelomeric enhancers.
Nucleic Acids Res, 50: 11580-11599 (2022) - ※Iwasaki YW, et al.
Piwi–piRNA complexes induce stepwise changes in nuclear architecture at target loci.
EMBOJ, 40: e108345 (2021) - Hasuwa H, Iwasaki YW. et al.
Production of functional oocytes requires maternally expressed PIWI genes and piRNAs in golden hamsters.
Nat Cell Biol, 23: 1002-1012 (2021) - #Murano K,#※Iwasaki YW. (#equally contributed) et al.
Nuclear RNA export factor variant initiates piRNA-guided co-transcriptional silencing.
EMBOJ, 38: e102870 (2019)
坂下 陽彦
- Otsuka K, et al., Sakashita A.
KRAB-zinc-finger proteins regulate endogenous retroviruses to sculpt germline transcriptome and genome evolution.
Genome Res, 35: 417-431 (2025) - ※Sakashita A, et al.
Polycomb protein SCML2 mediates paternal epigenetic inheritance through sperm chromatin.
Nucleic Acids Res, 51: 6668-6683 (2023) - Sakashita A, et al.
Transcription of MERVL retrotransposons is required for preimplantation embryo development.
Nat Genet, 55: 484-495 (2023) - ※Sakashita A, et al.
Bioinformatics pipelines for identification of super-enhancers and 3D chromatin contacts.
Methods Mol Biol (Epigenomics), 2577: 123-146 (2023) - Sakashita A, et al.
Endogenous retroviruses drive species-specific germline transcriptomes in mammals.
Nat Struct Mol Biol, 27: 967-977 (2020)
